12 Ocena jakości estymacji
library('gstat')
library('sp')
library('ggplot2')
library('caret')
library('geostatbook')
data(punkty)
data(siatka)12.1 Statystyki jakości estymacji
12.1.1 Statystyki jakości estymacji
W momencie, gdy trzeba określić jakość estymacji lub porównać wyniki pomiędzy estymacjami należy zastosować tzw. statystyki jakości estymacji. Do podstawowych statystyk ocen jakości estymacji należą:
- Średni błąd predykcji (MPE, ang. mean prediction error)
- Pierwiastek średniego błędu kwadratowego (RMSE, ang. root square prediction error)
- Rozkład błędu (np. 5. percentyl, mediana, 95. percentyl)
Idealna estymacja dawałaby brak błędu oraz współczynnik korelacji pomiędzy pomiarami (całą populacją) i szacunkiem równy 1. Wysokie, pojedyncze wartości błędu mogą świadczyć, np. o wystąpieniu wartości odstających.
12.1.2 Średni błąd estymacji
Średni błąd estymacji (MPE) można wyliczyć korzystając z poniższego wzoru:
\[ MPE=\frac{\sum_{i=1}^{n}(\hat{v}_i-v_i)}{n} \]
, lub używając funkcji mean() w R.
MPE <- mean(estymacja - obserwowane)Optymalnie wartość średniego błędu estymacji powinna być jak najbliżej 0.
12.1.3 Pierwiastek średniego błędu kwadratowego
Pierwiastek średniego błędu kwadratowego (RMSE) jest możliwy do wyliczenia poprzez wzór:
\[ RMSE=\sqrt{\frac{\sum_{i=1}^{n}(v_i-\hat{v}_i)^2}{n}} \]
, jak i proste obliczenie w R.
RMSE <- sqrt(mean((obserwowane - estymacja)^2))Optymalnie wartość pierwiastka średniego błędu kwadratowego powinna być jak najmniejsza.
12.1.4 Statystyki jakości estymacji | Mapa
Do oceny przestrzennej jakości predykcji można również zastosować mapę przestawiającą błędy predykcji (błędy estymacji). Wyliczenie błędów estymacji odbywa się poprzez odjęcie od predykcji wartości obserwowanej.
blad_predykcji <- estymacja - obserwowane## [using simple kriging]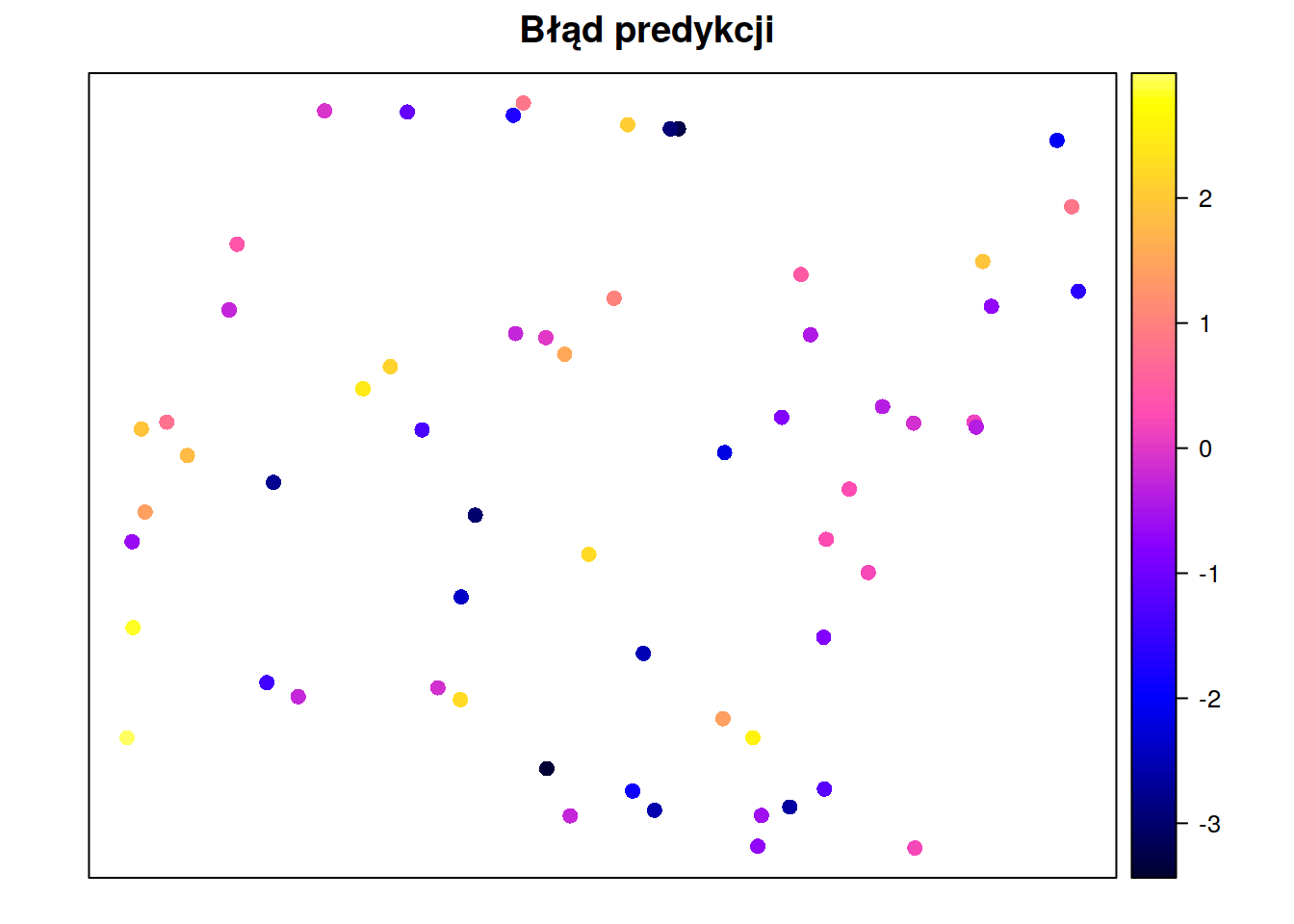
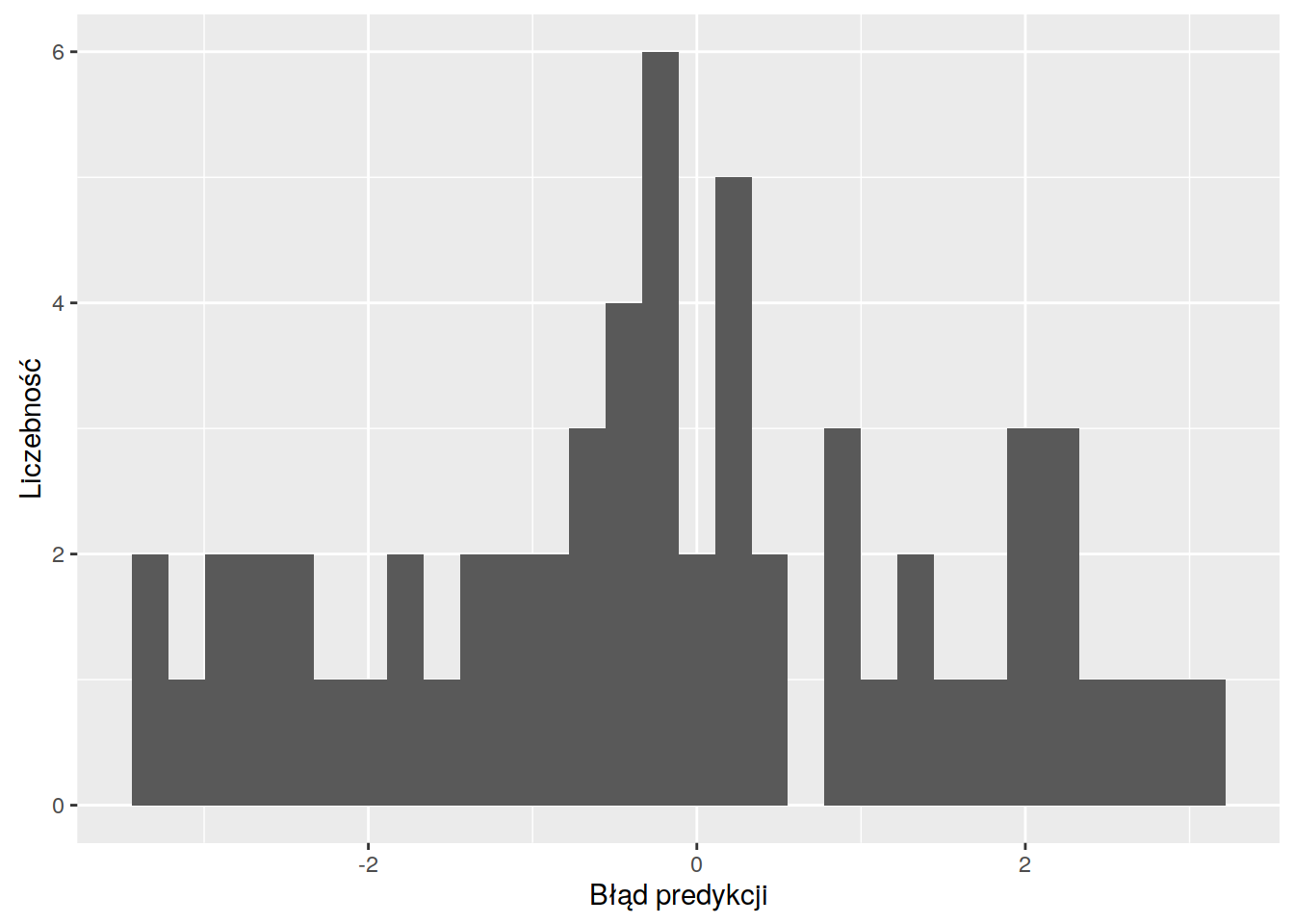
12.1.5 Statystyki jakości estymacji | Histogram
Błąd predykcji można również przedstawić na wykresach, między innymi, na histogramie.
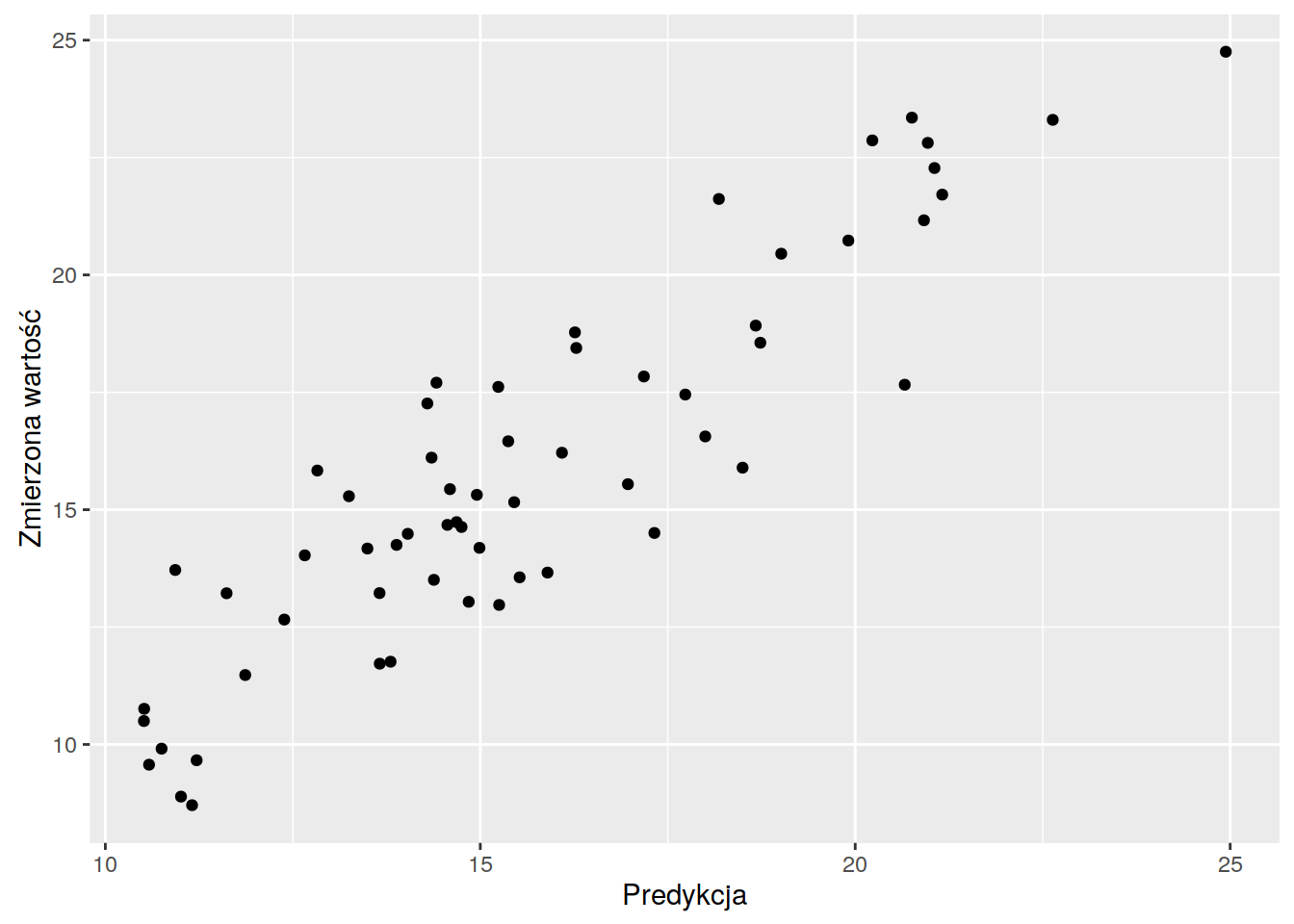
12.1.6 Statystyki jakości estymacji | Wykres rozrzutu
Do porównania pomiędzy wartością estymowaną a obserwowaną może również posłużyć wykres rozrzutu.
12.2 Walidacja wyników estymacji
12.2.1 Walidacja wyników estymacji
Dokładne dopasowanie modelu do danych może w efekcie nie dawać najlepszych wyników. Szczególnie będzie to widoczne w przypadku modelowania, w którym dane obarczone są znacznym szumem (zawierają wyraźny błąd) lub też posiadają kilka wartości odstających. W efekcie ważne jest stosowanie metod pozwalających na wybranie optymalnego modelu. Do takich metod należy, między innymi, walidacja podzbiorem (ang. jackknifing) oraz kroswalidacja (ang. crossvalidation).
12.2.2 Walidacja podzbiorem
Walidacja podzbiorem polega na podziale zbioru danych na dwa podzbiory - treningowy i testowy. Zbiór treningowy służy do stworzenia semiwariogramu empirycznego, zbudowania modelu oraz estymacji wartości. Następnie wynik estymacji porównywany jest z rzeczywistymi wartościami ze zbioru testowego. Zaletą tego podejścia jest stosowanie danych niezależnych od estymacji do oceny jakości modelu. Wadą natomiast jest konieczność posiadania (relatywnie) dużego zbioru danych.
Na poniższym przykładzie zbiór danych dzielony jest używając funkcji createDataPartition() z pakietu caret. Użycie tej funkcji powoduje stworzenie indeksu zawierającego numery wierszy dla zbioru treningowego. Ważną zaletą funkcji createDataPartition() jest to, iż w zbiorze treningowym i testowym zachowane są podobne rozkłady wartości. W przykładzie użyto argumentu p=0.75, który oznacza, że 75% danych będzie należało do zbioru treningowego, a 25% do zbioru testowego. Następnie korzystając ze stworzonego indeksu, budowane są dwa zbiory danych - treningowy (train) oraz testowy (test).
set.seed(124)
indeks <- as.vector(createDataPartition(punkty$temp, p=0.75, list=FALSE))
indeks## [1] 1 2 4 7 8 9 12 13 14 15 16 17 18 19 22 23 25 26 27 29
## [21] 30 32 34 35 36 37 38 40 41 42 43 45 49 51 52 53 54 55 56 58
## [41] 59 60 61 62 64 66 67 69 70 73 75 76 78 79 81 82 83 84 85 86
## [61] 87 88 89 90 91 94 96 97 98 100 101 102 103 106 107 108 109 110 111 112
## [81] 113 114 115 117 118 120 122 124 125 127 128 129 130 132 133 134 135 137 138 139
## [101] 140 141 142 143 144 146 148 149 150 153 154 155 156 157 158 159 160 161 162 163
## [121] 167 168 169 171 173 174 175 176 177 180 181 183 184 185 186 187 188 189 190 191
## [141] 193 194 195 196 199 200 201 202 204 205 208 209 210 212 213 214 215 216 217 218
## [161] 219 220 221 223 225 226 227 228 229 230 231 232 233 234 235 236 237 238 239 240
## [181] 241 242 243 244 245 246 247 248 249 250train <- punkty[indeks, ]
test <- punkty[-indeks, ]Dalszym krokiem jest stworzenie semiwariogramu empirycznego oraz jego modelowanie w oparciu o zbiór treningowy.
vario <- variogram(temp~1, data=train)
model <- vgm(10, model = 'Sph', range = 4000, nugget = 0.5)
fitted <- fit.variogram(vario, model)
plot(vario, model=fitted)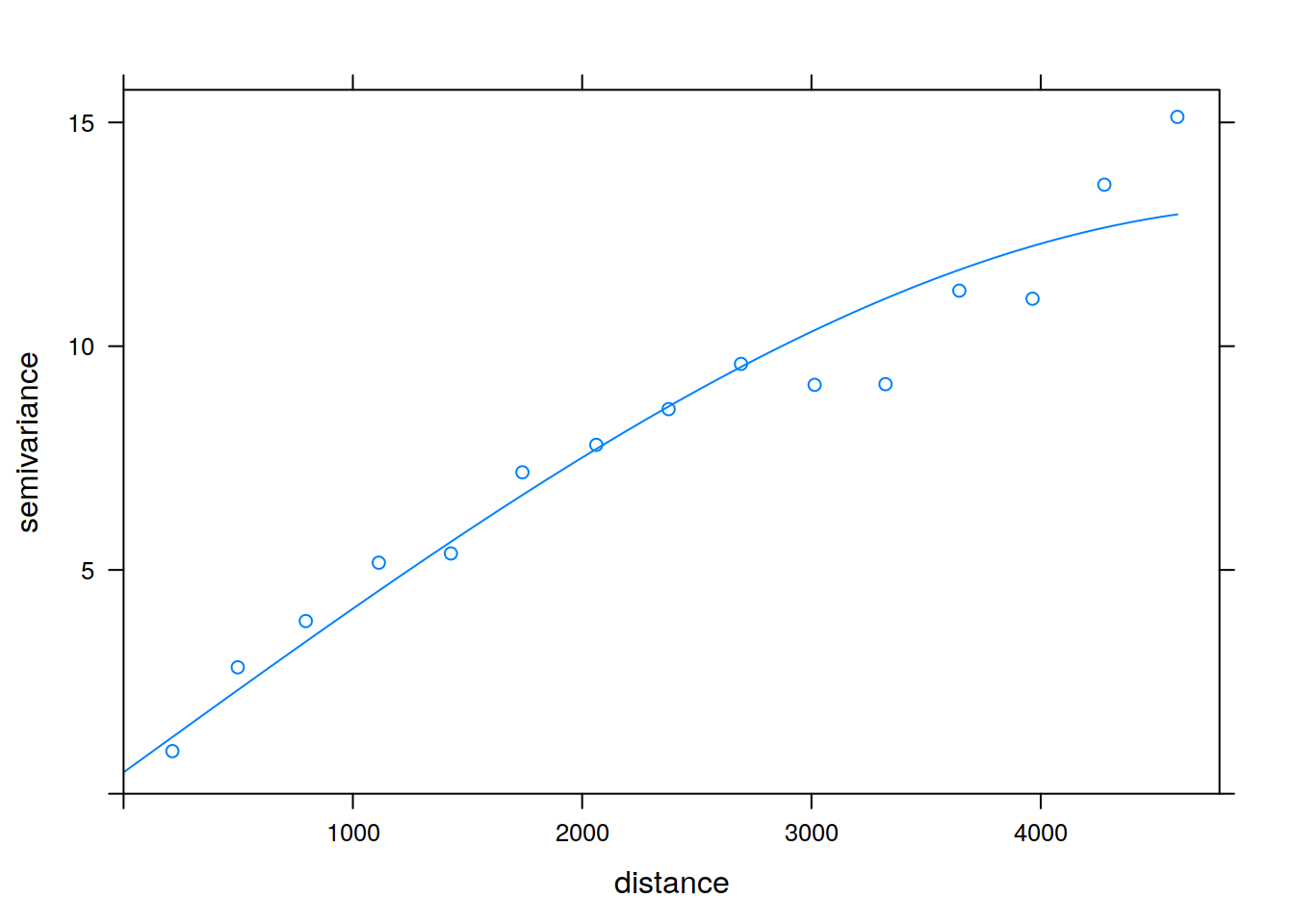
Do porównania wyników estymacji w stosunku do zbioru testowego posłuży funkcja krige(). Wcześniej wymagała ona podania wzoru, zbioru punktowego, siatki oraz modelu. W tym przypadku jednak chcemy porównać wynik estymacji i testowy zbiór punktowy. Dlatego też, zamiast obiektu siatki definiujemy obiekt zawierający zbiór testowy (test).
test_sk <- krige(temp~1, train, test, model=fitted, beta=15.324)## [using simple kriging]summary(test_sk)## Object of class SpatialPointsDataFrame
## Coordinates:
## min max
## x 745731 756567.0
## y 712629 721118.7
## Is projected: TRUE
## proj4string :
## [+init=epsg:2180 +proj=tmerc +lat_0=0 +lon_0=19 +k=0.9993 +x_0=500000
## +y_0=-5300000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs]
## Number of points: 60
## Data attributes:
## var1.pred var1.var
## Min. :10.51 Min. :1.002
## 1st Qu.:13.62 1st Qu.:1.654
## Median :14.97 Median :2.084
## Mean :15.66 Mean :2.187
## 3rd Qu.:18.05 3rd Qu.:2.608
## Max. :24.94 Max. :5.602Uzyskane w ten sposób wyniki możemy określić używając statystyk jakości estymacji lub też wykresów.
blad_predykcji_sk <- test_sk$var1.pred - test$temp
summary(blad_predykcji_sk)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -3.4360 -1.3860 -0.2442 -0.1954 0.9098 2.9990MPE <- mean(test_sk$var1.pred - test$temp)
MPE## [1] -0.1953769RMSE <- sqrt(mean((test$temp-test_sk$var1.pred)^2))
RMSE## [1] 1.692512test_sk$blad_predykcji_sk <- blad_predykcji_sk
spplot(test_sk, 'blad_predykcji_sk')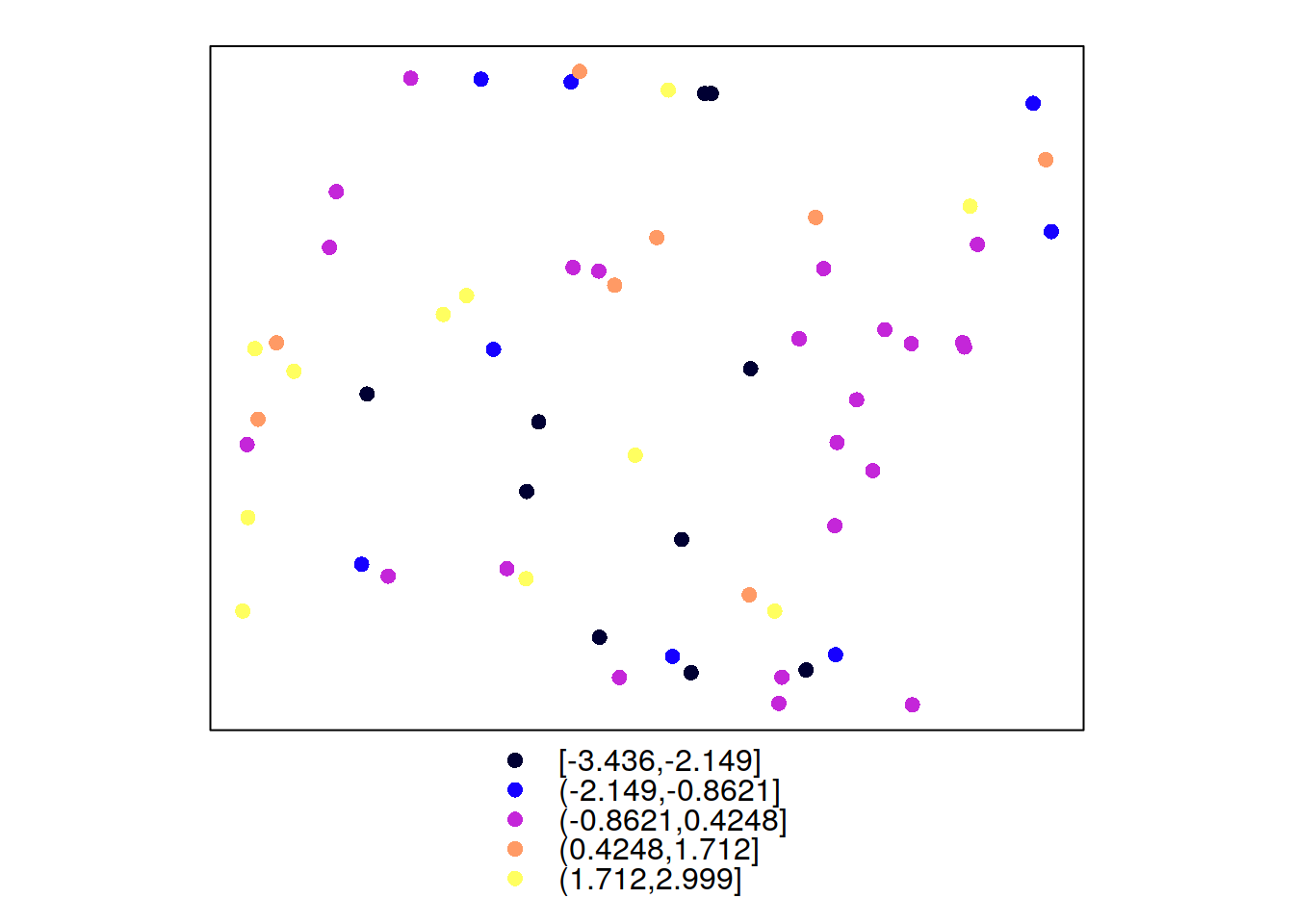
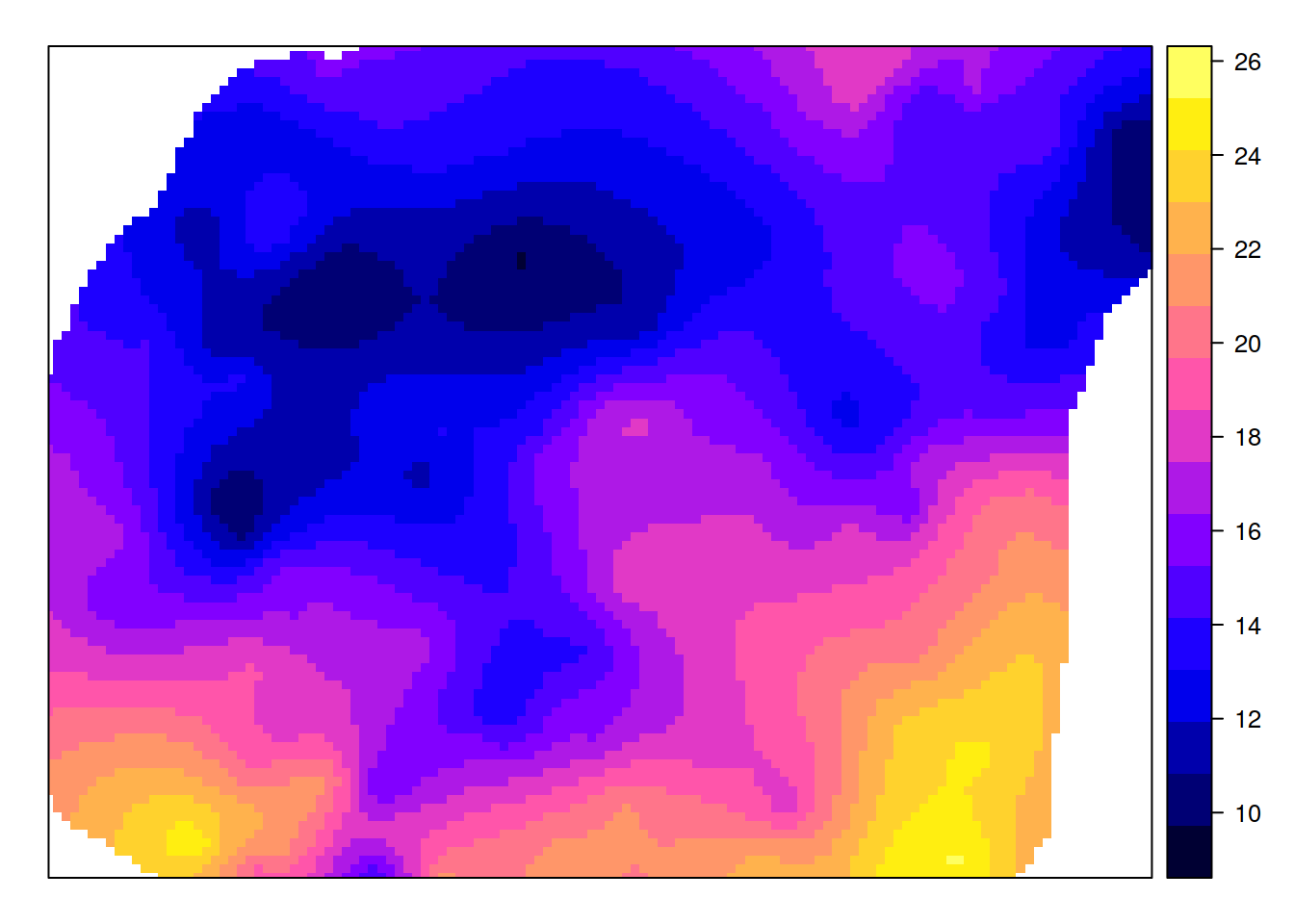
W sytuacji, gdy uzyskany model jest wystarczająco dobry, możemy również uzyskać estymację dla całego obszaru z użyciem funkcji krige(), tym razem jednak podając obiekt siatki.
test_sk <- krige(temp~1, train, siatka, model=fitted, beta=15.324)## [using simple kriging]spplot(test_sk, 'var1.pred')
12.2.3 Kroswalidacja
W przypadku kroswalidacji te same dane wykorzystywane są do budowy modelu, estymacji, a następnie do oceny prognozy. Procedura kroswalidacji LOO (ang. leave-one-out cross-validation) składa się z poniższych kroków:
- Zbudowanie matematycznego modelu z dostępnych obserwacji
- Dla każdej znanej obserwacji następuje:
- Usunięcie jej ze zbioru danych
- Użycie modelu do wykonania predykcji w miejscu tej obserwacji
- Wyliczenie reszty (ang. residual), czyli różnicy pomiędzy znaną wartością a estymacją
- Podsumowanie otrzymanych wyników
W pakiecie gstat, kroswalidacja LOO jest dostępna w funkcjach krige.cv() oraz gstat.cv(). Działają one bardzo podobnie jak funkcje krige() oraz gstat(), jednak w przeciwieństwie do nich nie wymagają podania obiektu siatki.
vario <- variogram(temp~1, data=punkty)
model <- vgm(10, model = 'Sph', range = 4000, nugget = 0.5)
fitted <- fit.variogram(vario, model)
cv_sk <- krige.cv(temp~1, punkty, model=fitted, beta=15.324, verbose=FALSE)
summary(cv_sk)## Object of class SpatialPointsDataFrame
## Coordinates:
## min max
## x 745546.9 756937.4
## y 712618.8 721192.6
## Is projected: NA
## proj4string : [NA]
## Number of points: 250
## Data attributes:
## var1.pred var1.var observed residual
## Min. : 9.786 Min. :1.219 Min. : 8.706 Min. :-8.96908
## 1st Qu.:13.333 1st Qu.:1.806 1st Qu.:13.284 1st Qu.:-0.95234
## Median :15.298 Median :2.093 Median :15.309 Median : 0.04980
## Mean :15.936 Mean :2.220 Mean :15.950 Mean : 0.01423
## 3rd Qu.:18.062 3rd Qu.:2.477 3rd Qu.:18.273 3rd Qu.: 0.85669
## Max. :24.994 Max. :5.320 Max. :26.139 Max. : 6.45468
## zscore fold
## Min. :-4.930132 Min. : 1.00
## 1st Qu.:-0.647920 1st Qu.: 63.25
## Median : 0.036433 Median :125.50
## Mean : 0.008082 Mean :125.50
## 3rd Qu.: 0.598859 3rd Qu.:187.75
## Max. : 3.330852 Max. :250.00spplot(cv_sk, 'residual')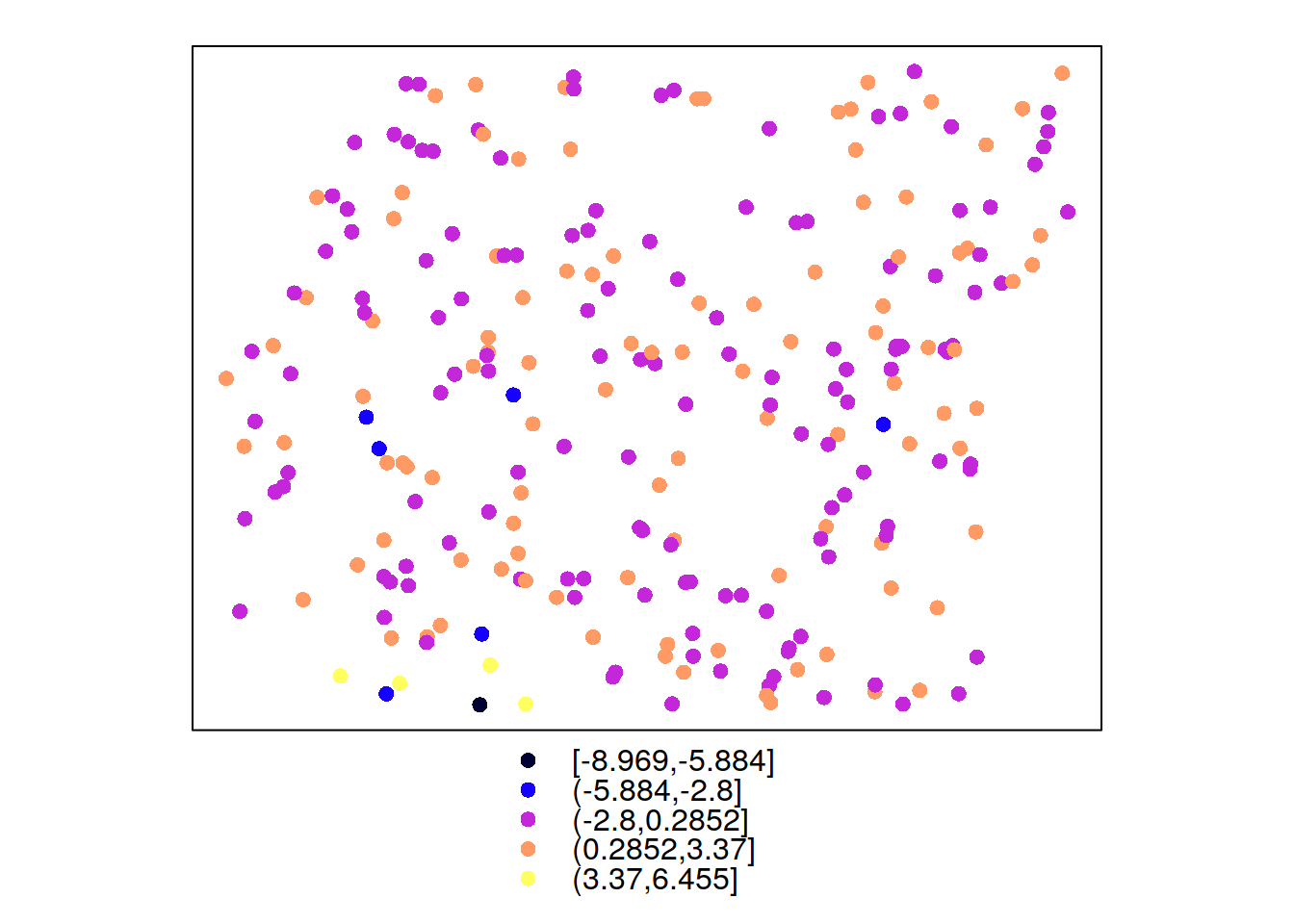
Podobnie jak w walidacji podzbiorem, gdy uzyskany model jest wystarczająco dobry, estymację dla całego obszaru uzyskuje się z użyciem funkcji krige().
cv_skk <- krige(temp~1, train, siatka, model=fitted, beta=15.324)## [using simple kriging]spplot(cv_skk, 'var1.pred')