10 Estymacje wielozmienne
library('gstat')
library('sp')
library('geostatbook')
data(punkty)
data(punkty_ndvi)
data(siatka)10.1 Kokriging
10.1.1 Kokriging (ang. co-kriging)
Kokriging pozwala na wykorzystanie dodatkowej zmiennej (ang. auxiliary variable), zwanej inaczej kozmienną (ang. co-variable), która może być użyta do prognozowania wartości badanej zmiennej w nieopróbowanej lokalizacji. Zmienna dodatkowa może być pomierzona w tych samych miejscach, gdzie badana zmienna, jak też w innych niż badana zmienna. Możliwa jest też sytuacja, gdy zmienna dodatkowa jest pomierzona w dwóch powyższych przypadkach. Kokriging wymaga, aby obie zmienne były istotnie ze sobą skorelowane. Najczęściej kokriging jest stosowany w sytuacji, gdy zmienna dodatkowa jest łatwiejsza (tańsza) do pomierzenia niż zmienna główna. W efekcie, uzyskany zbiór danych zawiera informacje o badanej zmiennej oraz gęściej opróbowane informacje o zmiennej dodatkowej. Jeżeli informacje o zmiennej dodatkowej są znane dla całego obszaru wówczas bardziej odpowiednią techniką będzie kriging z zewnętrznym trendem (KED).
10.1.2 Kokriging | Wybór dodatkowej zmiennej
Wybór zmiennej dodatkowej może opierać się na dwóch kryteriach:
- Teoretycznym
- Empirycznym
10.2 Krossemiwariogramy
10.2.1 Krossemiwariogramy
Metoda kokrigingu opiera się nie o semiwariogram, lecz o krossemiwariogramy. Krossemiwariogram jest to wariancja różnicy pomiędzy dwiema zmiennymi w dwóch lokalizacjach. Wyliczając krossemiwariogram otrzymujemy empiryczne semiwariogramy dla dwóch badanych zmiennych oraz kroswariogram dla kombinacji dwóch zmiennych.
W poniższym przykładzie istnieją dwie zmienne, savi ze zbioru punkty pomierzona w 250 lokalizacjach oraz ndvi ze zbioru punkty_ndvi pomierzona w 997 punktach.
spplot(punkty, 'savi')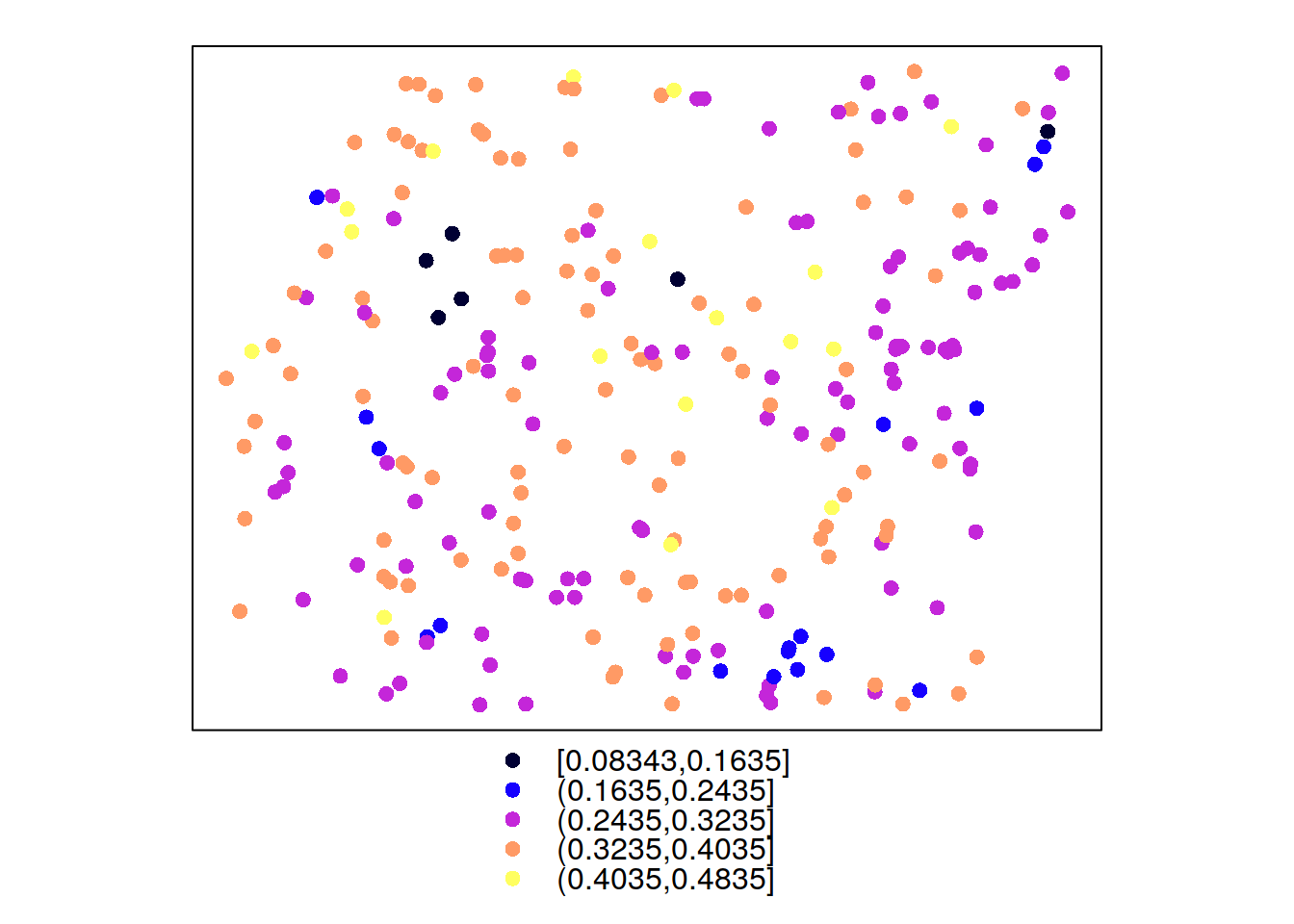
spplot(punkty_ndvi, 'ndvi')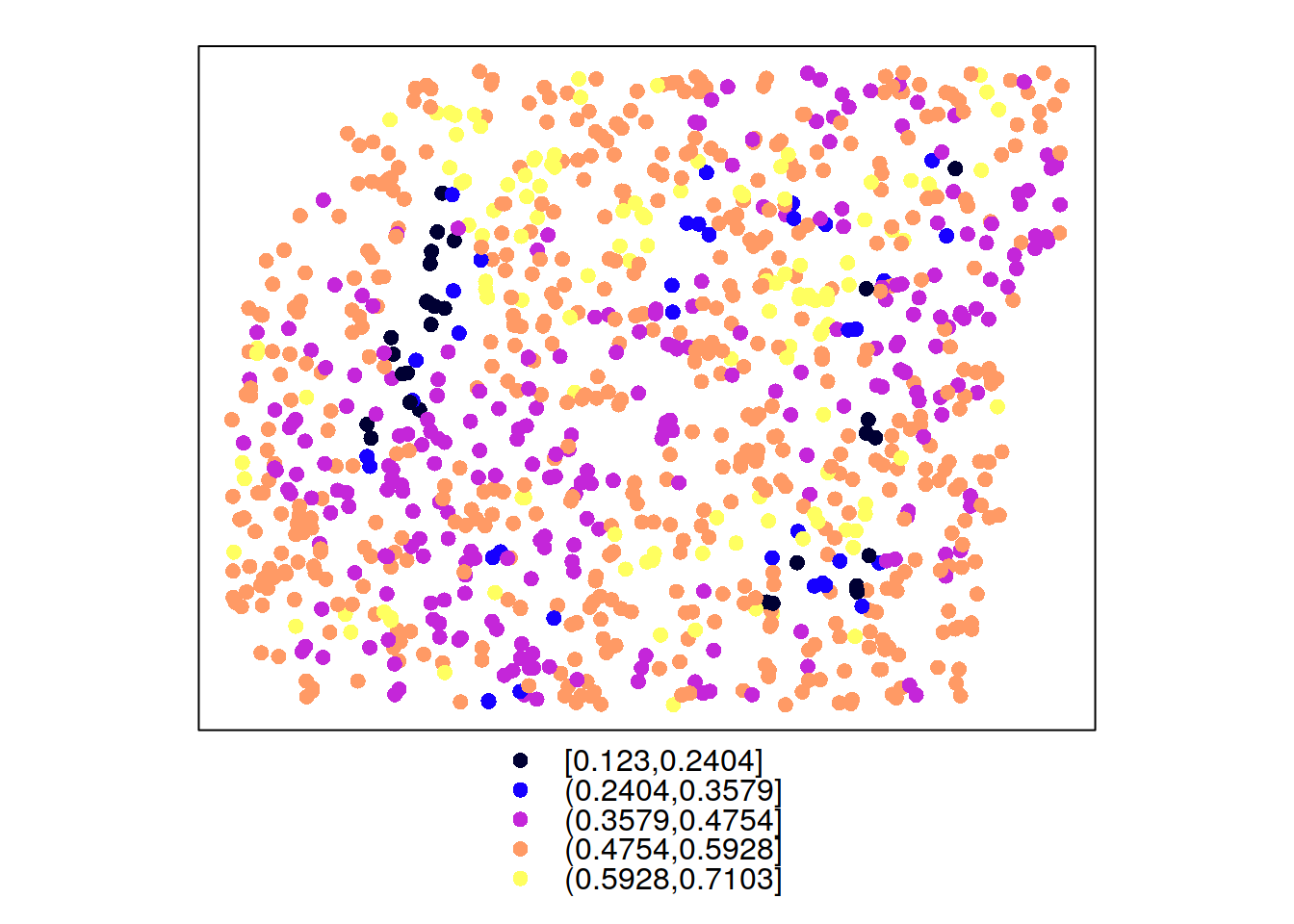
Tworzenie krossemiwariogramów odbywa się z użyciem funkcji gstat(). Na początku definiujemy pierwszy obiekt g. Składa się on z obiektu pustego (NULL), nazwy pierwszej zmiennej (nazwa może być dowolna), wzoru (w przykładzie savi~1), oraz pierwszego zbioru punktowego. Następnie do pierwszego obiektu g dodajemy nowe informacje również poprzez funkcję gstat(). Jest to nazwa obiektu (g), nazwa drugiej zmiennej, wzór, oraz drugi zbiór punktowy.
g <- gstat(NULL, id='SAVI', form = savi~1, data = punkty)
g <- gstat(g, id='NDVI', form = ndvi~1, data = punkty_ndvi)
g## data:
## SAVI : formula = savi`~`1 ; data dim = 250 x 5
## NDVI : formula = ndvi`~`1 ; data dim = 997 x 1Z uzyskanego w ten sposób obiektu tworzymy krossemiwariogram (funkcja variogram()), a następnie go wizualizujemy używając funkcji plot().
v <- variogram(g)
plot(v)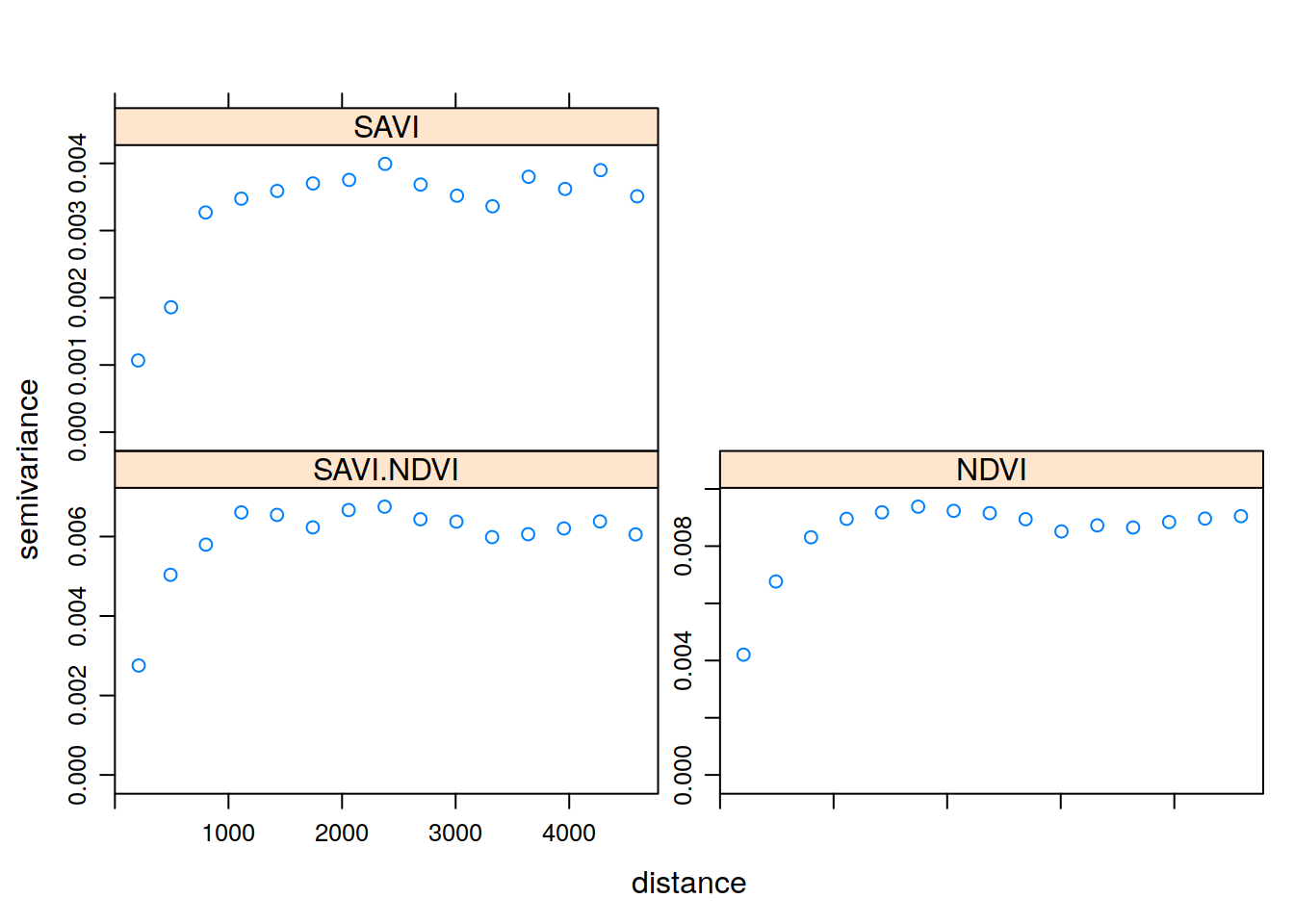
10.3 Modelowanie krossemiwariogramów
10.3.1 Modelowanie krossemiwariogramów
Modelowanie krossemiwariogramów, podobnie jak ich tworzenie, odbywa się używając funkcji gstat(). Podaje się w niej wcześniejszy obiekt g, model, oraz argument fill.all=TRUE. Ten ostatni parametr powoduje, że model dodawany jest do wszystkich elementów krossemiwariogramu.
g <- gstat(g, model=vgm(0.006, 'Sph', 2000, 0.001), fill.all=TRUE)W przypadku semiwariogramów funkcja fit.variogram() służyła dopasowaniu parametrów modelu do semiwariogramu empirycznego. Podobną rolę w krossemiwariogramach spełnia funkcja fit.lmc(). Dopasowuje ona liniowy model koregionalizacji do semiwariogramów wielozmienych. Funkcja fit.lmc() oczekuje co najmniej dwóch elementów, krossemiwariogramu oraz modelów krossemiwariancji. W poniższym przykładzie dodatkowo użyto parametru correct.diagonal = 1.01, z uwagi na to że analizowane zmienne wykazywały bardzo silną korelację.
# zmienne są bardzo mocno skorelowane - dlatego użyto argumentu correct.diagonal = 1.01
g_fit <- fit.lmc(v, g, correct.diagonal = 1.01)
g_fit## data:
## SAVI : formula = savi`~`1 ; data dim = 250 x 5
## NDVI : formula = ndvi`~`1 ; data dim = 997 x 1
## variograms:
## model psill range
## SAVI[1] Nug 0.001278509 0
## SAVI[2] Sph 0.003338946 2000
## NDVI[1] Nug 0.004065525 0
## NDVI[2] Sph 0.005918453 2000
## SAVI.NDVI[1] Nug 0.002257298 0
## SAVI.NDVI[2] Sph 0.004352821 2000plot(v, g_fit)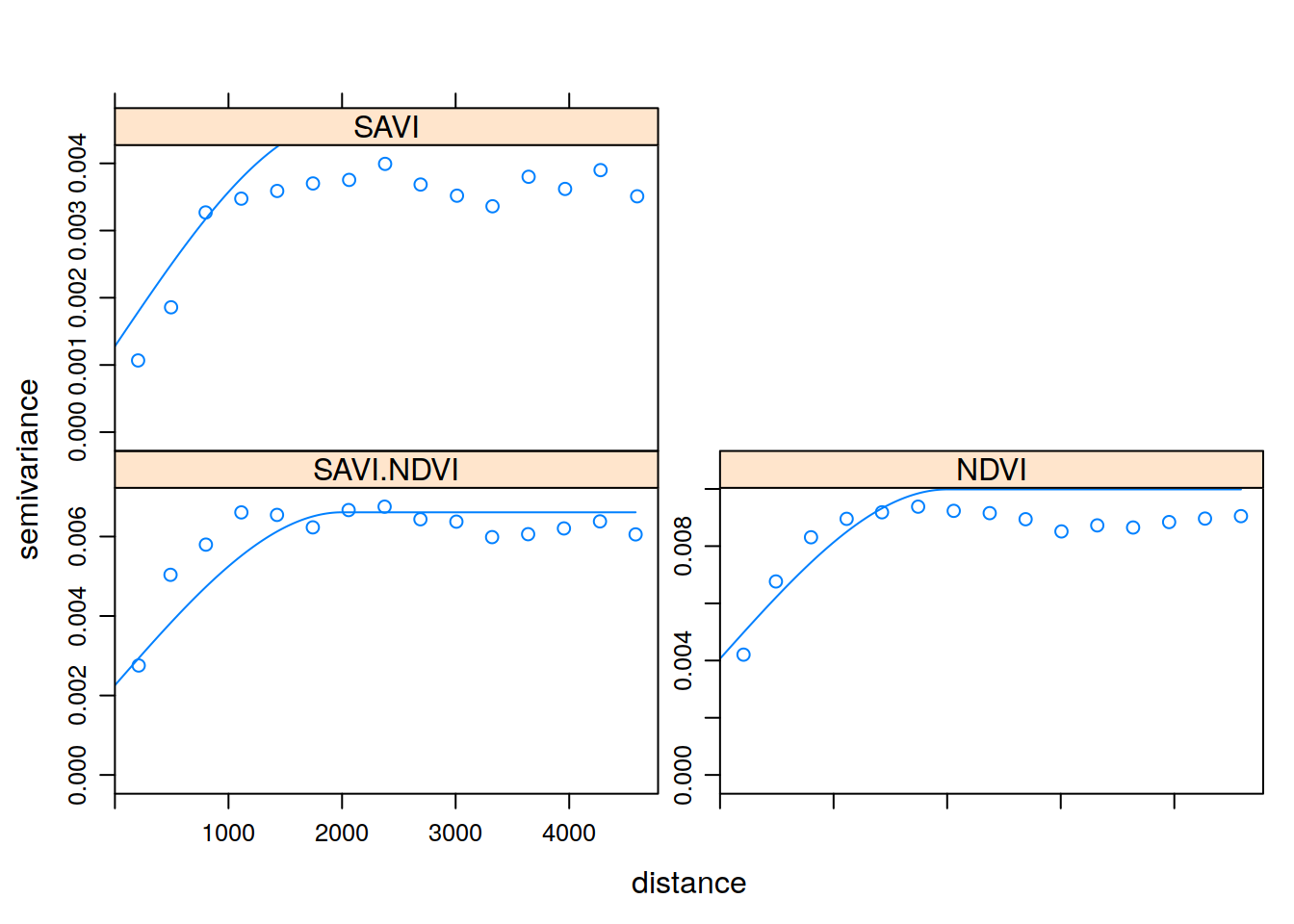
10.4 Kokriging
10.4.1 Kokriging
Posiadając dopasowane modele oraz siatkę można uzyskać wynik używając funkcji predict().
ck <- predict(g_fit, siatka) ## Linear Model of Coregionalization found. Good.
## [using ordinary cokriging]W efekcie otrzymujemy pięć zmiennych:
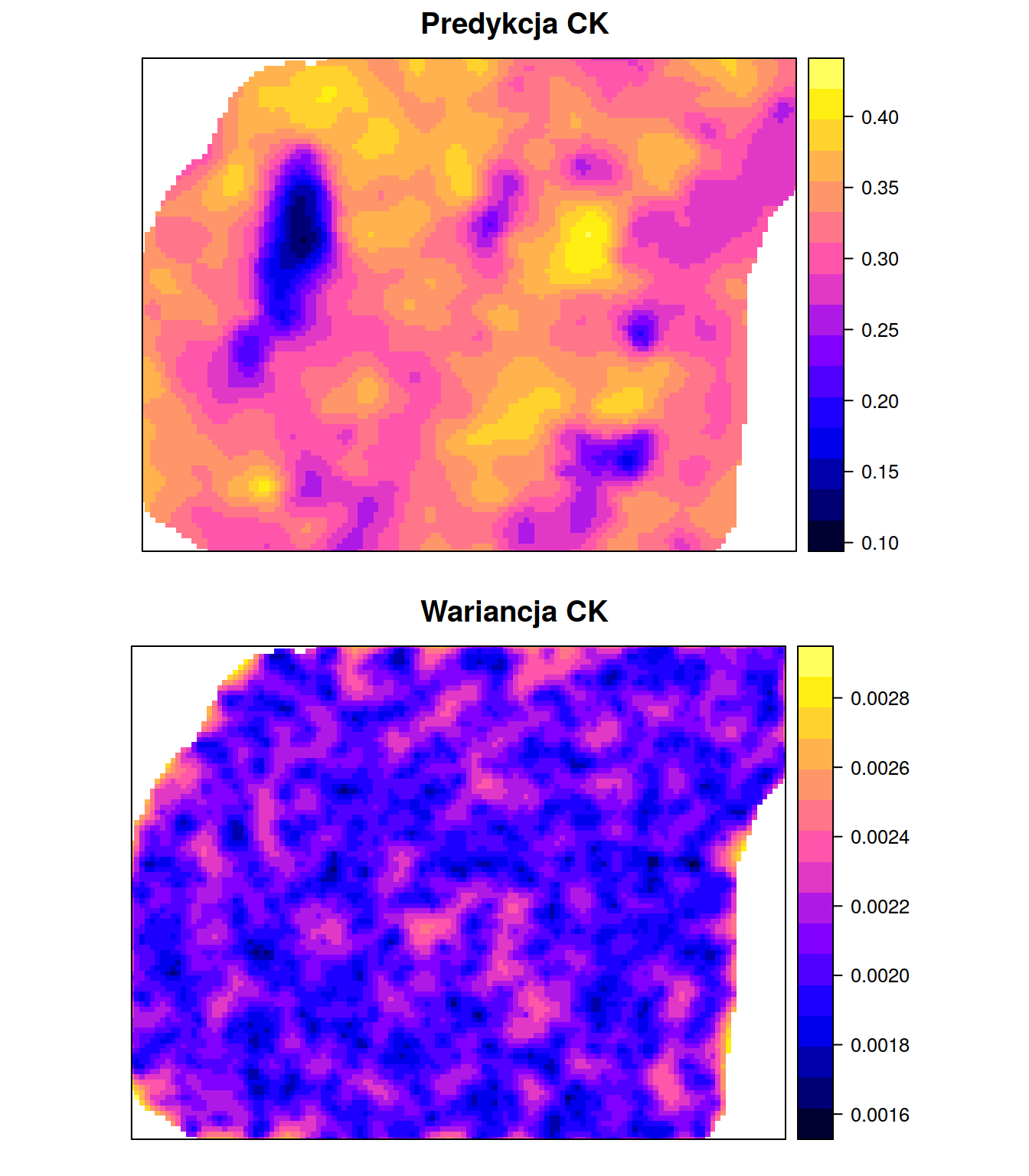
SAVI.pred- estymacja zmiennejsaviSAVI.var- wariancja zmiennejsaviNDVI.pred- estymacja zmiennejndviNDVI.var- wariancje zmiennejndvicov.SAVI.NDVI- kowariancje zmiennychsaviorazndvi
summary(ck)## Object of class SpatialPixelsDataFrame
## Coordinates:
## min max
## x 745586.7 756926.7
## y 712661.2 721211.2
## Is projected: TRUE
## proj4string :
## [+init=epsg:2180 +proj=tmerc +lat_0=0 +lon_0=19 +k=0.9993 +x_0=500000
## +y_0=-5300000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs]
## Number of points: 10993
## Grid attributes:
## cellcentre.offset cellsize cells.dim
## s1 745586.7 90 127
## s2 712661.2 90 96
## Data attributes:
## SAVI.pred SAVI.var NDVI.pred NDVI.var
## Min. :0.1152 Min. :0.001615 Min. :0.2336 Min. :0.004750
## 1st Qu.:0.3001 1st Qu.:0.001958 1st Qu.:0.4715 1st Qu.:0.005188
## Median :0.3255 Median :0.002046 Median :0.5056 Median :0.005336
## Mean :0.3205 Mean :0.002060 Mean :0.4996 Mean :0.005365
## 3rd Qu.:0.3480 3rd Qu.:0.002149 3rd Qu.:0.5365 3rd Qu.:0.005513
## Max. :0.4197 Max. :0.002862 Max. :0.6280 Max. :0.006780
## cov.SAVI.NDVI
## Min. :0.002658
## 1st Qu.:0.003045
## Median :0.003157
## Mean :0.003178
## 3rd Qu.:0.003289
## Max. :0.004232spplot(ck, 'SAVI.pred')
spplot(ck, 'SAVI.var')